DigiSkin Atlas
Eine offene Ressource für RNA-Sequenzdaten der Haut.
RNA-Sequenzdaten geben Einblick darin, welche Gene aktuell in der Haut abgeschrieben und umgesetzt werden. So können Eigenschaften und Verhalten von Zellen charakterisiert werden. Derzeit müssen diese Daten in jedem Labor immer wieder neu generiert werden, da Daten aus bereits veröffentlichten Studien bruchstückhaft und für andere Forscher oft unzugänglich sind. Das wollen wir ändern.
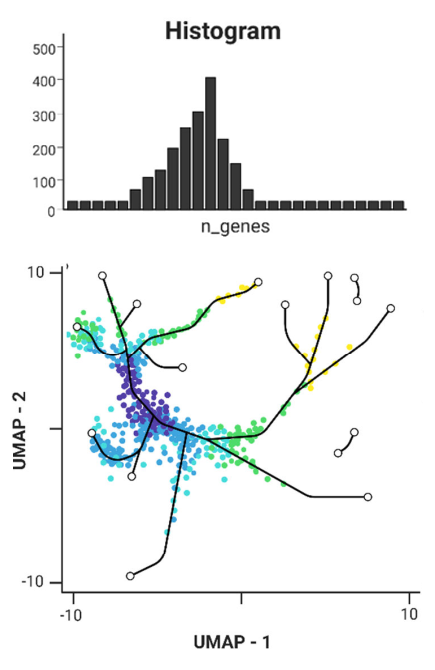
Aus 20 veröffentlichten Studien mit Single Cell Sequencing Daten der Maushaut wurde bereits ein robuster Datensatz erstellt. Wir liefern einen Proof-of-Concept-Beweis dafür, dass der Datensatz die Erkennung kleiner Zellpopulationen wie seneszente Zellen ermöglicht. Darüber hinaus überprüfen wir die Konsistenz der In-silico-Analyse mit bereits veröffentlichten Ergebnissen und In-vivo-Analysen.
Unser Ziel ist es nun, diese Plattform zu erweitern, indem wir weitere Daten von verschiedenen Genotypen, Hautkrankheiten und Hautanhangsgebilden integrieren. In Zusammenarbeit mit Expert:innen wird ein digitaler Hautatlas erschaffen, der nicht nur die Notwendigkeit von Tiemodellen reduziert, sondern auch bahnbrechende Einblicke in die Hautbiologie und zelluläre Seneszenz ermöglicht. Der DigiSkin-Atlas soll online zugänglich gemacht werden, um Wissenschaftler:innen eine leistungsstarke Ressource zur Erforschung verschiedener Erkrankungen und Phänotypen in der Maushaut zu bieten.
In unserem Labor werden wir den Datensatz verwenden, um die Merkmale der zellulären Seneszenz in Bezug auf die mit der Wundheilung verbundenen Faktoren wie Epithel-zu-Mesenchym-Übergangsmarker, migrationsfördernde Faktoren, Zytokine und andere zu analysieren. Dies wird an verschiedenen Hautzelltypen, einschließlich Keratinozyten, Fibroblasten und Immunzellen, durchgeführt und durch histologische In-situ-Experimente verifiziert.
Das Projekt wird ermöglicht durch eine 3R/Stand-Alone Förderung des FWF (Grant-DOI: 10.55776/P37321). Wir bedanken uns herzlich für die Unterstützung.